文:回溯档案
编辑:回溯档案
盐度等非生物胁迫越来越严重地威胁着作物生产力并降低了全球植物的质量,盐度会影响细胞渗透压和离子稳态以及光合作用;耗尽细胞能量;并导致氧化还原失衡、生长减缓,甚至植物死亡。
植物必须通过操纵专门的基因表达程序来形成有效的策略来适应各种生物和非生物胁迫,以促进胁迫保护、体内平衡和生存,近十年来,越来越多的研究集中在拟南芥和水稻等模式植物耐盐性的分子机制上。

了解这些模式植物的耐盐机制无疑对于研究这些作物的耐盐机制具有重要意义。
棉花是一种重要的经济作物,是第二大固有耐盐作物,比水稻和拟南芥更耐盐,然而当棉株暴露在高盐度条件下时,尤其是在发芽或幼苗阶段时,会出现生长抑制和生产力下降。

而且随着耕地面积的减少,粮棉竞争日益突出,因此棉花种植逐渐向盐碱地转移,因此为了选育耐盐棉花品种,阐明棉花的耐盐机制并鉴定耐盐相关基因至关重要。
不幸的是由于驯化后期对产量的选择,大多数棉花品种的耐盐性逐渐丧失,因为了鉴定耐盐基因/蛋白质,优选分析具有相似遗传背景和对比耐盐性的基因型。

事实上,最近将糖生植物拟南芥的盐响应与盐生植物的盐响应进行了比较,除了这些研究之外,其他涉及水稻的研究、小麦、番茄、棉花 和大豆进行了研究,以了解盐敏感和耐盐基因型之间基因/蛋白质表达的差异。
尽管对耐盐胁迫相关的分子调控途径有广泛的描述,但涉及“组学”的大规模实验过程促进了对棉花耐盐机制中基因型相关差异的解释和蛋白质组学。

蛋白质组的精确分析对于理解潜在的应激生理学至关重要,并且需要在多个层面上进一步阐明。
尽管最近进行了研究,但由于转录过程中mRNA剪接的复杂性,对蛋白质组的完整描述仍然具有挑战性,除了转录调控外,基因表达在转录后水平也受到明显的调控。

在我们的研究中,通过利用同量异序标签进行基于相对和绝对定量(iTRAQ)的蛋白质组学(既高通量又定量),以及同一样品中mRNA、选择性剪接(AS)和miRNA的转录组数据。
我们第一次能够深入分析这些重要参与者的基因表达之间的关系,并揭示两种对比基因型在盐胁迫期间的基因调控机制,我们比较了蛋白质和mRNA的表达水平,并分析了mRNA和蛋白质丰度差异之间的相关性。

结果表明,基因和蛋白表达谱相关性较弱,但一些应激反应基因根据mRNA水平和翻译效率进行调节,这对于应激诱导基因和应激抑制基因都很明显。
一些基因表现出相反的趋势,表明拮抗调节发生在mRNA和翻译水平,我们的分析结果还揭示了不同的耐盐机制,即棉花代谢和应激反应途径的几个新方面存在差异,揭示它们与AS事件和miRNA的关系受到调节。

这项研究为线粒体内耐盐相关基因/蛋白质与能量代谢之间的相互关系提供了新线索,棉花代谢和应激反应途径的几个新方面有所不同,揭示它们与AS事件和miRNA的关系受到调节。
这项研究为线粒体内耐盐相关基因/蛋白质与能量代谢之间的相互关系提供了新线索,棉花代谢和应激反应途径的几个新方面有所不同,揭示它们与AS事件和miRNA的关系受到调节。

这项研究为线粒体内耐盐相关基因/蛋白质与能量代谢之间的相互关系提供了新线索。
植物材料和盐胁迫处理
两种陆地棉基因型的植物,即耐盐的和对盐敏感的南丹巴迪大花(NH),在含有1/2强度Hoagland的水培容器中种植植物加速器中的溶液。
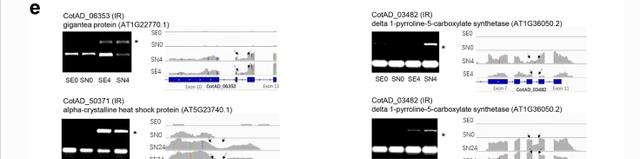
暴露于200mMNaCl溶液4或24小时后,收获对照和处理过的NH和E7幼苗的叶子并直接浸入液氮中,然后将其储存在-80°C下直至提取mRNA和蛋白质。
从每个基因型/处理中取样超过六片幼苗叶子,每个样品进行三次生物学重复以进行蛋白质组分析;在这些重复中,其中一个还用于转录组测序(mRNA-seq和小RNA-seq),如Peng先前所述。

又一年在与上述相同的条件下独立种植新批次的幼苗并生长,收集了更严格质量的幼苗样本,以进一步验证转录组数据。
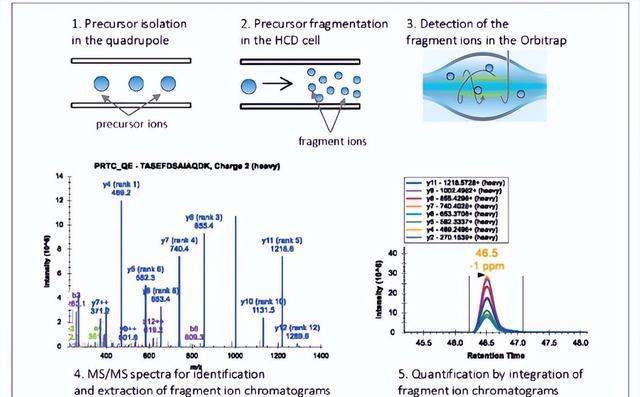
蛋白质提取、iTRAQ标记和LC-ESI质谱(MS)/MS分析
如前所述,分离和纯化经过200mMNaCl处理0、4或24小时的幼苗的总蛋白,将18个样品(即2个基因型×3个时间点×3个重复)的总蛋白上清液转移至新管中,并使用2-D定量试剂盒(美国通用电气公司)进行定量。

进行了三次生物学重复和三次技术重复以验证蛋白质质量和浓度。iTRAQ分析由华大基因进行。
从每个样品溶液中除去总蛋白质(约100μg)含量,然后用胰蛋白酶消化蛋白质,随后通过真空离心干燥肽,将肽在0.5M四乙基溴化铵(TEAB)中重构,并使用iTRAQ8-plex试剂盒,根据制造商的说明进行标记。
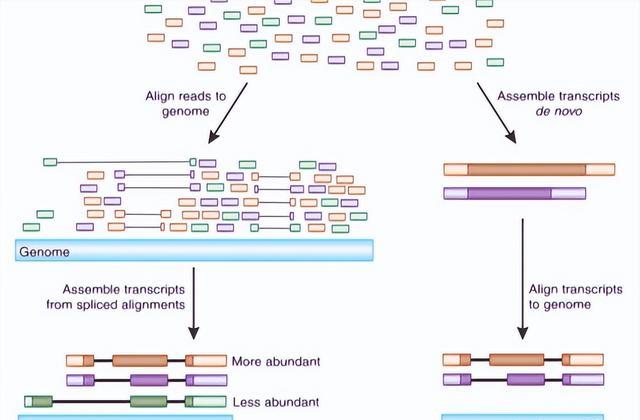
转录组测序和AS分析
所有六个cDNA(用于mRNA-seq)文库和所有六个原始数据的小RNA文库均从NCBI序列读取存档下载登记号SRP043419,重要的是,转录组和蛋白质组数据源自同一批次的样品。
去除低质量序列读数后,使用干净的读数映射到陆地棉AD 1基因组和参考序列,具有95%的最低同一性,所有六个文库中的基因表达均标准化为每百万个映射读数中每千碱基外显子模型的读数(RPKM)。

如果一个基因的|log则被认为是两个样本之间的差异表达(DE)2比例| ≥1且FDR小于0.001,同时,TopHat(2.1.0版)被用来预测各种类型的流行AS事件。
此外我们将经历AS的差异表达基因(DEG)定义为选择性剪接DEG(AS-DEG)。

盐胁迫下mRNA和蛋白质的不同表达模式
使用iTRAQ技术对六个样本进行定量蛋白质组分析,mRNA-seq和小RNA-seq监测转录组变化,显示了本研究中使用的数据分析策略的概述。使用陆地棉蛋白质数据库时的蛋白质组结果,包括肽谱匹配质量(质量增量)、数量、分布、蛋白质序列覆盖度和每次重复的数量。
总体而言,在三个生物重复中总共鉴定了4004个非冗余蛋白质,使用至少两种肽并进行两次重复鉴定了2810种蛋白质的子集。
为了比较这些蛋白质的丰度及其相对于各自基因转录水平的丰度变化,使用相同的样品进行了相应的mRNA-seq实验,在盐胁迫存在和/或不存在的情况下,4小时和24小时后,在两种棉花基因型的叶子中总共检测到42,234个表达基因。
定量全局蛋白质组分析与转录组分析同时进行,本质上揭示了在不同水平观察到的不同表达模式,在全球范围内,根据SE4/SE0、SE24/SE0、SN4/SN0和SN24/SN0的蛋白质和基因表达谱确定了六个不同的类别。
分析了蛋白质组和mRNA-seq结果之间所有常见基因/蛋白质的相关性,以研究蛋白质积累在哪个水平上受到调节,结果表明,4小时(r?=0.034)和24小时(r=0.038)。

然而DAP与其相应的mRNA之间观察到更强的正相关或负相关;这些相关性相同或与变化方向相反(r =0.9035,0.8208(4h)。
III类和IV类,mRNA或蛋白质水平的DE
我们的蛋白质组和mRNA-seq分析结果显示,502(4小时)和587(24小时)基因是DE,但其相应的蛋白质保持不变(III类),此外在盐胁迫下,4小时和24小时的48和92个蛋白质在蛋白质水平上是DE,而不是在mRNA水平上(IV类)。
并分别在盐胁迫4小时和24小时后在蛋白质水平上进行了定量,毫不奇怪,这些蛋白质的丰度与其相应的AS-DEG之间没有检测到显着差异。
这些结果表明,尽管一些盐响应基因的表达受到AS事件的影响,但其相应蛋白质的丰度在盐胁迫条件下保持不变,盐胁迫4小时后基于GO的基因功能注释表明。

各种生物过程的调节和关联描绘了陆地棉的耐盐信号传导模式
在这里我们首先使用基于iTRAQ的定量蛋白质组学方法来分析两种对比棉花基因型的六个棉花样品中的蛋白质,这些蛋白质代表盐胁迫的两个关键阶段。
然后我们将这些蛋白质的表达与其mRNA水平进行比较,确定了几种潜在的基因调控机制,我们发现转录和转录后调控可能与AS和miRNA的产生有关。

在我们的工作之前,已经进行了几项涉及盐处理棉苗的叶、茎和根的转录组学和蛋白质组学研究,这些研究在RNA和蛋白质水平上积累了大量关于耐盐分子机制的信息。
通过利用基于iTRAQ的常见样本的蛋白质组、转录组、AS和miRNA数据,我们首次能够深入分析基因表达中这些重要参与者之间的关系,并揭示盐胁迫期间基因调控的几种机制。

我们确定了一些生物过程,例如“磷脂酰肌醇信号系统”、“色氨酸代谢”、“磷酸肌醇代谢”以及“乙醛酸和二羧酸代谢”,这些过程可能较少受到耐盐额外调节机制的影响。
然而对于大多数强DAP,没有观察到其相应mRNA水平的显着变化,同样对于大多数DEG,没有观察到其相应蛋白质水平的显着变化,另一方面,一些蛋白质在应激条件下可能并不迫切需要,必须在mRNA水平上合成并在短时间内翻译以发挥其功能。

结论
我们全面分析了两种不同的耐盐棉花基因型响应盐胁迫的mRNA、miRNA和蛋白质谱的整体变化,总体而言使用iTRAQ比率对2316种蛋白质进行了定量。
在所有三个生物重复中对1090种蛋白质进行了定量,在存在和/或不存在盐胁迫的情况下,4小时和24小时后,在两种棉花基因型的叶子中总共检测到42,234个表达基因。
额外分析的结果显示,在蛋白质组的4004个蛋白质中,检测到了3162个基因/蛋白质,蛋白质组和mRNA-seq数据之间的关联分析结果显示,一些基因在蛋白质组和mRNA水平上都是DE,但我们也发现大多数强DAP在其相应的mRNA水平上没有显着变化。
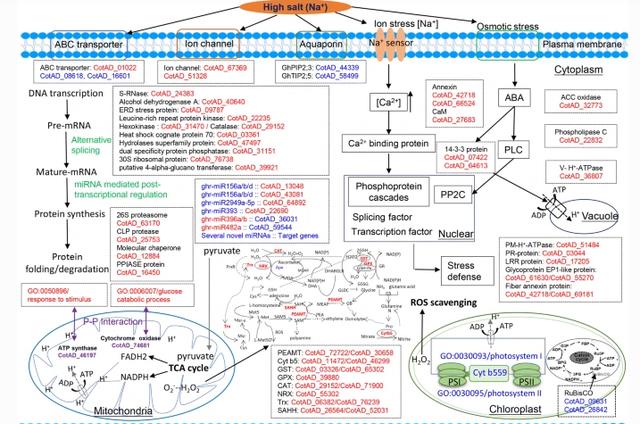
最后,我们预测了一个包含158个基因/蛋白质的相互作用网络,然后发现了线粒体中由ATP合酶(CotAD_74681)和细胞色素氧化酶(CotAD_46197)组成的两个主要簇。
总的来说,我们为系统耐盐模型提供了一个可能的示意图;该示意图揭示了棉花响应盐胁迫的多个水平的基因调控。
参考文献
【1】MunnsR,TesterM。盐度耐受机制。植物生物学年鉴。2008;59:651–81。
【2】朱建坤. 植物的耐盐性。植物科学趋势。2001;6:66-71。
【3】测试员M,达文波特R。高等植物中的Na +耐受性和Na +运输。安·博特. 2003;91:503-27。
【4】花TJ。提高作物的耐盐性。J实验机器人。2004;55:307。
【5】MunnsR.基因和耐盐性:将它们结合在一起。新植醇。2005;167:645。
