
引言
有性生殖过程中的同源重组,增加了自然选择的遗传多样性,并能去除有害的突变。性别二态性普遍存在于真核生物中,通常由基因组中的性别决定区(sex-determining region, SDR)控制。为了维持性别的保真性,SDR进化出了广泛的重组抑制,这种抑制在众多物种中由不同机制调控,包括但不限于序列差异、结构重排、组蛋白修饰等等【1】。
莱茵衣藻(Chlamydomonas reinhardtii)作为一种单倍体的单细胞真核绿藻,是研究有性生殖与减数分裂的重要模式生物。衣藻的性别由mating-type locus (mt) 决定,有两种交配型:plus与minus。MT是基因组中一段375 kb (mt+) 或211 kb (mt–) 的复杂序列【2】,在两种交配型之间重排。此前的研究表明,重排序列对该区域的重组抑制有所贡献,但并不充分【3】,同时两者的因果关系也并不明晰。
从进化的角度来看,DNA早于染色体出现,意味着DNA修饰的出现可能同样早于组蛋白修饰。DNA胞嘧啶甲基化(5mC)是真核生物中常见的表观遗传修饰,在多个生物学过程中发挥着重要作用。衣藻作为进化早期兼具动植物特性的生物,其性别决定区MT的高甲基化赋予了性别维持机制新的可能。
2024年10月9日,中国科学院分子细胞科学卓越创新中心徐国良研究员、中国科学院上海营养与健康研究所邵振研究员、分子细胞科学卓越创新中心陈辉副研究员在Science Advances发表文章:DNA cytosine methylation suppresses meiotic recombination at the sex-determining region,该研究揭示了DNA甲基化在维持性别决定区稳定性中的重要作用。
 作者首先鉴定了衣藻中负责细胞核DNA甲基化的胞嘧啶甲基转移酶,确认DNMT1 (Cre10.g461750) 是主要的维持性甲基转移酶,并通过实验室前期开发的CRISPR技术【4】得到了多株dnmt1突变体。通过四分体分析,作者发现在减数分裂过程中,DNMT1的完全缺失会导致子代生存率的降低以及生存谱式的异常,并在MT发生非正常重组从而产生mixed-mt的异常子代。通过对重组频率的检测与统计,作者发现相较于WT × WT对照组,dnmt1 × dnmt1组别的确在高甲基化的MT发生相当比例的重组(对照组中频率为0);而在常染色体区域,两组的重组频率并无差异。ChIP-seq结果表明,dnmt1突变藻株中,MT区域的H3K9me1修饰显著降低。为了探究H3K9me1的降低与表型之间的关系,作者进一步构建了SET3(衣藻中产生H3K9me1修饰的主要甲基转移酶)敲除藻株,发现该基因的突变恰好能模拟dnmt1突变藻株中H3K9me1的降低(总量与分布),同时并未对5mC水平造成影响。但在四分体分析检测中,set3 × set3组别并无异常表型产生。这说明衣藻中5mC与H3K9me1是严格的上下游关系,组蛋白修饰的改变只是DNA甲基化改变的结果,并非产生表型的原因。衣藻中的发现让我们有理由推测,真核生物从20亿年前进化至今,DNA修饰或许行使了更多功能。研究DNA甲基化在其他物种(如哺乳动物)中,是否贡献于性别决定区的重组抑制,将对探索表观遗传修饰的进化具有重要意义。
作者首先鉴定了衣藻中负责细胞核DNA甲基化的胞嘧啶甲基转移酶,确认DNMT1 (Cre10.g461750) 是主要的维持性甲基转移酶,并通过实验室前期开发的CRISPR技术【4】得到了多株dnmt1突变体。通过四分体分析,作者发现在减数分裂过程中,DNMT1的完全缺失会导致子代生存率的降低以及生存谱式的异常,并在MT发生非正常重组从而产生mixed-mt的异常子代。通过对重组频率的检测与统计,作者发现相较于WT × WT对照组,dnmt1 × dnmt1组别的确在高甲基化的MT发生相当比例的重组(对照组中频率为0);而在常染色体区域,两组的重组频率并无差异。ChIP-seq结果表明,dnmt1突变藻株中,MT区域的H3K9me1修饰显著降低。为了探究H3K9me1的降低与表型之间的关系,作者进一步构建了SET3(衣藻中产生H3K9me1修饰的主要甲基转移酶)敲除藻株,发现该基因的突变恰好能模拟dnmt1突变藻株中H3K9me1的降低(总量与分布),同时并未对5mC水平造成影响。但在四分体分析检测中,set3 × set3组别并无异常表型产生。这说明衣藻中5mC与H3K9me1是严格的上下游关系,组蛋白修饰的改变只是DNA甲基化改变的结果,并非产生表型的原因。衣藻中的发现让我们有理由推测,真核生物从20亿年前进化至今,DNA修饰或许行使了更多功能。研究DNA甲基化在其他物种(如哺乳动物)中,是否贡献于性别决定区的重组抑制,将对探索表观遗传修饰的进化具有重要意义。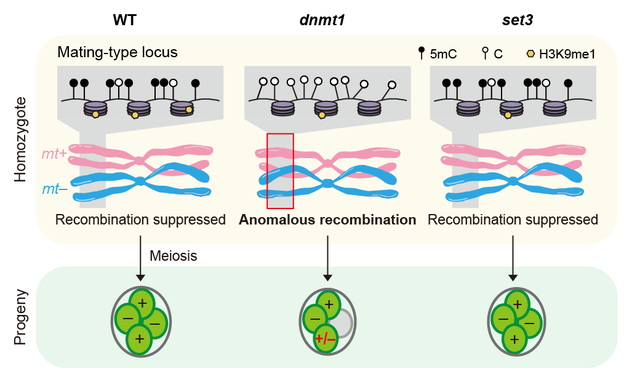
模式图(Credit: Science Advances)
综上所述,该研究发现了莱茵衣藻中DNA甲基化对交配型决定区重组抑制的重要功能,揭示了性别决定区维持的新机制,拓展了DNA甲基化的生物学功能。参考文献
1. B. L. S. Furman, D. C. H. Metzger, I. Darolti, A. E. Wright, B. A. Sandkam, P. Almeida, J. J. Shu, J. E. Mank, Sex Chromosome Evolution: So Many Exceptions to the Rules. Genome Biol Evol 12, 750-763 (2020).2. U. Goodenough, H. Lin, J. H. Lee, Sex determination in Chlamydomonas. Semin Cell Dev Biol 18, 350-361 (2007).3. D. Lopez, T. Hamaji, J. Kropat, P. De Hoff, M. Morselli, L. Rubbi, S. Fitz-Gibbon, S. D. Gallaher, S. S. Merchant, J. Umen, M. Pellegrini, Dynamic Changes in the Transcriptome and Methylome of Chlamydomonas reinhardtii throughout Its Life Cycle. Plant Physiol 169, 2730-2743 (2015).4. H. Chen, Q. L. Yang, J. X. Xu, X. Deng, Y. J. Zhang, T. Liu, M. G. Rots, G. L. Xu, K. Y. Huang, Efficient methods for multiple types of precise gene-editing in Chlamydomonas. Plant J 115, 846-865 (2023).https://www.science.org/doi/epdf/10.1126/sciadv.adr2345责编|探索君
排版|探索君
文章来源|“BioArt”
End
